의료에서의 Whole Slide Image와 인공지능
🧫 병리학(Pathology)에서의 Whole Slide Image(WSI)가 무엇인지 알아보고, 전처리 방법과 관련 AI 연구 동향을 공부해봅시다.
KEYWORDS
의료 AI, Medical AI, CPath, Whole Slide Image, Foundation Model, 의료 딥러닝, Cytopathology, Histopathology, Digital Pathology
Whole Slide Image(WSI)란?
- 의료 영역에서 Whole Slide Imaging 기술은 전통적인 유리 슬라이드를 디지털 이미지로 변환하는 기술이며, 병리 샘플을 스캐너를 통해 스캔하여 WSI를 생성하고 파일로 저장합니다.
- 병리학자(Pathologist)들은 WSI를 확대하여 특정 세포들의 구조를 자세히 관찰하고, 각 병변 영역이나 WSI 전체에서의 진단을 내립니다.
- WSI는 수십억 개의 픽셀을 포함하고 있는 고해상도 이미지이기에 파일 크기는 1GB에서 4GB정도로 큽니다.
- 이러한 거대 이미지는 일반적인 AI 모델로 쉽게 처리할 수 없기 때문에, 새로운 접근 방식이 필요합니다.
- 대표적으로
.mrxs,.svs,.ndpi,.tiff등의 확장자를 따릅니다.- 이러한 형식은 이미지를 만드는 데 사용된 스캐너에 따라 다릅니다.
- ex. Aperio, NanoZoomer, Pannoramic
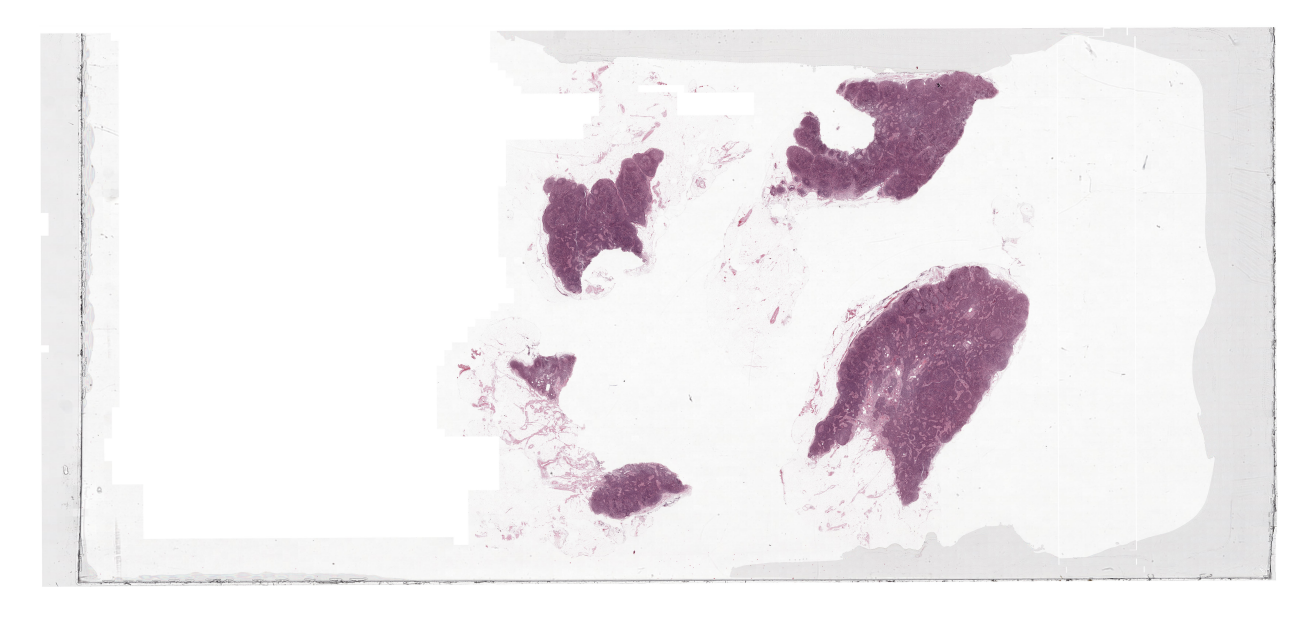 A whole slide image of histopathology 1
A whole slide image of histopathology 1
WSI 불러오기
- WSI는 일반적으로 다중 계층 피라미드 형식으로 저장되어 있으며, 다양한 해상도의 이미지 레벨에 접근할 수 있습니다.
- 예를 들어, 피라미드
.tiff형식의 WSI의 경우, 각 해상도는 파일 내에서 별도의 레이어 또는 페이지로 저장되는 경우가 많습니다.- 페이지 0에는 WSI의 전체 해상도 이미지가 들어있으며, 이후의 페이지에는 더 낮은 해상도의 이미지가 들어가게 됩니다.
- 보통 각 레이어는 이전의 레이어를 2배 다운샘플링(Downsampling)한 것으로 구성됩니다.
- 각 레이어는 \(512 \times 512\) 크기의 타일로 저장되며, JPEG와 같은 형식으로 압축됩니다.
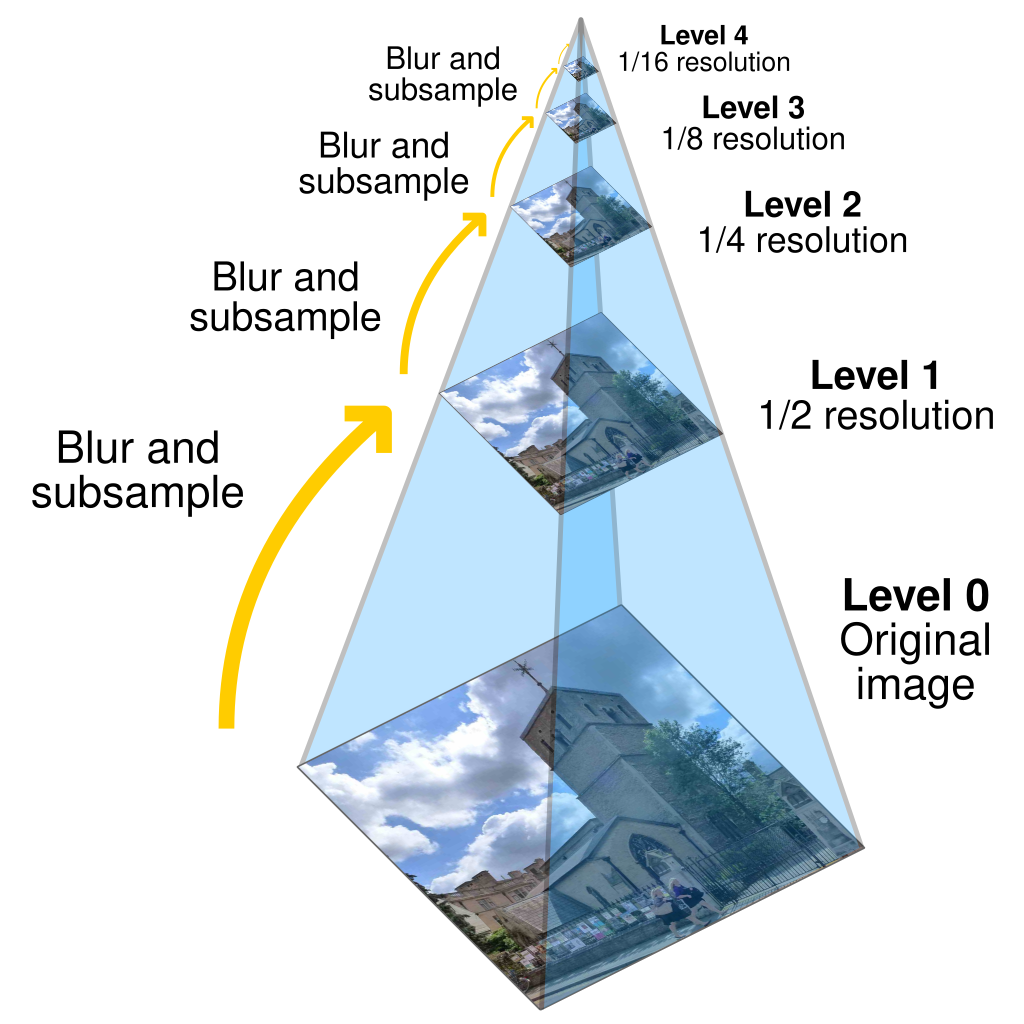 Pyramid structure of the whole slide image 2
Pyramid structure of the whole slide image 2
- OpenSlide 라이브러리
- C기반 라이브러리로, WSI를 읽기 위해 설계되었습니다.
- Python 바인딩이 포함되어 있어 Deep Zoom 생성과 같은 추가 기능이 제공됩니다.
- 기능
- WSI 읽기 ㅣ 슬라이드 이미지를 객체형태로 불러옵니다.
- 크기 읽기 ㅣ 각 해상도 계층의 크기 정보에 접근합니다.
- 영역 읽기 ㅣ 원하는 해상도 수준에서 WSI의 특정 영역을 추출합니다.
- 타일 추출 ㅣ 지정된 크기로 WSI를 작은 패치(타일)로 나눌 수 있어 패치 기반 분석에 유용합니다.
1
2
3
4
5
6
7
8
9
10
!pip install openslide-python
import openslide
slide = openslide.OpenSlide("slide.tiff")
slide_shape = slide.dimensions
slide_region = slide.read_region((0, 0), 0, (256, 256))
slide_region.show()
print(slide_shape)
(70000, 70000, 3)
- PyVips 라이브러리
- 큰 이미지 작업에 활용되는 라이브러리인 libvips에 대한 Python 바인딩입니다.
- 파일을 한 번에 메모리에 불러오지 않고, 필요한 부분만 읽어오는 특징이 있어 OOM 문제가 잘 발생하지 않습니다.
- 타일 추출과 같은 기본적인 기능이 모두 빠르게 작동하며, Numpy와의 호환성이 좋습니다.
- 내부적으로 멀티스레딩을 지원하므로 많은 WSI 데이터를 한번에 분석하는 작업에서 유리합니다.
1
2
3
4
5
6
7
8
9
!pip install pyvips
import pyvips
slide = pyvips.Image.new_from_file("slide.tiff")
slide = np.ndarray(buffer=slide.write_to_memory(), dtype=np.uint8, shape=[slide.height, slide.width, slide.bands])
slide = slide[:, :, :3]
print(slide.shape)
(70000, 70000, 3)
WSI 전처리하기
- 패치 추출(Patch Extraction)
- 고해상도 WSI의 전체 크기를 다루는 대신, 이미지를 더 작은, AI를 통해 처리 가능한 단위인 정사각형 패치(Patch)로 나누어 분석합니다.
- 이러한 패치 크기는 보통 \(256 \times 256\) 픽셀에서 \(1024 \times 1024\) 픽셀까지 다양하게 설정할 수 있습니다.
- 핵심 정보 손실 없이 효율적인 처리와 분석을 가능하게 합니다.
- 다운샘플링(Downsampling)
- WSI 해상도를 낮추어 전체 이미지를 처리할 수 있게 하는 방법이지만, 세부 이미지 정보가 손실될 위험이 있습니다.
- 따라서, 패치 기반 방법이 더 많이 사용됩니다.
패치 기반(Patch-level) WSI 분석
- 조직/세포 분할(Tissue/Cell Segmentation)
- 불필요한 영역(배경 또는 흐릿한 부분 등)을 식별하고 제거합니다.
- 배경 제거(Background Removal)
- 학습 데이터셋에는 유의미한 조직/세포 패치만 포함되도록 보장 되어야 합니다.
- 임계값 설정(Thresholding) ㅣ 배경인지 아닌지를 결정하는 고정된 임계값이 사용될 수 있습니다.
- ex. 패치 내 평균 픽셀 값이 임계값을 초과하면 해당 패치는 배경으로 간주할 수 있습니다.
- 의미 있는 조직/세포 데이터가 없는 불필요한 영역의 처리를 피하여 계산 부담을 줄입니다.
 A comparison of a whole slide image and the detection of white background areas 1
A comparison of a whole slide image and the detection of white background areas 1
- 색상 정규화(Colour Normalisation)
- 이미지 내 색상 값의 분포를 조정합니다.
- 여러 슬라이드 간에 색상 차이를 일관되게 유지하여, 학습 데이터 불균형을 줄이고 결과의 왜곡을 최소화합니다.
- 패치 추출(Patch Extraction)
- WSI를 \(256 \times 256\) 픽셀 크기의 정사각형 패치로 잘라냅니다.
- AI로 처리할 수 있는 단위로 많은 데이터를 재생산/저장할 수 있으며, 더 작은 영역에 집중하면서 빠르게 추론할 수 있습니다.
- 패치 크기, 해상도, 샘플링 방법, 중첩(Overlapping) 여부를 설정해야합니다.
- 중첩은 패치 추출 시 패치를 정의된 픽셀 수 만큼 겹치는 방법을 의미하며, 이는 패치 생성 시 손실될 수 있는 공간 정보를 보존하는 데 도움을 줍니다.
- 모든 패치를 생성한 후에는 특정 작업을 위해서 부분 집합을 선택하여 활용합니다.
- ex. 테스트 데이터는 전체 패치를 사용하고, 학습 데이터는 샘플만 사용합니다.
- 샘플링(Sampling)
- 랜덤 샘플링(Random Sampling) ㅣ 패치가 무작위로 선택됩니다.
- 클래스 불균형을 야기할 수 있습니다. (ex. 정상패치가 너무 많고 악성패치는 적은 경우)
- 각 패치는 중복 없이 선택되며, 이미지당 선택할 수 있는 최대 패치 수가 정해두는 경우가 많습니다.
- 정보적 샘플링(Informed Sampling) ㅣ 병변의 실제 위치를 고려하면서 패치를 선택합니다.
- 랜덤 샘플링(Random Sampling) ㅣ 패치가 무작위로 선택됩니다.
- 데이터 증강(Data Augmentation)
- 기존의 학습 데이터를 변형하여 새로운 학습 데이터를 생성합니다.
- 모델 학습 중 오버피팅을 방지하고, 클래스 불균형 문제를 해결하며 데이터의 변동성에 강한, Robust한 모델을 개발하는데 도움이 됩니다.
다운샘플링(Downsampling)
- WSI를 효율적으로 분석하기 위해 이미지 크기와 복잡성을 줄이는 과정입니다.
- 단독으로 사용하거나, 패치 추출 기법과 함께 사용할 수 있습니다.
- 다운샘플링이 충분히 이루어지면, 이미지를 패치로 나누지 않고도 AI 학습에 바로 활용할 수 있습니다.
- 패치 기반 방법 선호
- 일반적으로 다운샘플링은 중요한 세부정보, 특히 형태학적 정보의 손실을 일으킬 수 있어, 모델 예측의 정확도가 떨어질 수 있습니다.
AI로 WSI 분석하기
- WSI를 이용한 AI 모델 개발 분야에서, 딥러닝 모델은 병리학자가 어떻게 이미지를 분석하는지 모방하는 데 중점을 둡니다.
- CNN(Convolutional Neural Networks)은 고해상도 이미지 데이터에서 여러 병번과 세포에 대한 시각적인 패턴을 인식하는 데 효과적입니다.
- 그러나 이러한 모델을 학습시키기 위한 라벨이 부족하다는 문제가 있으며, 많은 연구자들은 패치 수준 주석(Patch-level Annotation)이나 약한 감독 학습(Weakly Supervised Learning)을 통해 이러한 문제를 해결하고 있습니다.
AI 모델 구조
- Patch Classifier + CNN(Convolutional Neural Network) ㅣ WSI와 같은 대형 이미지를 패치화하여 각 패치들에 대한 예측을 CNN 기반 모델을 통해 수행하는 방식입니다.
- MIL(Multiple Instance Learning) ㅣ 약한 지도 학습(Weakly Supervised Learning)에 속하는 방법이며, 슬라이드 전체에 대한 단일 라벨을 슬라이드의 여러 부분(패치)에 대한 분석을 종합(Patch Aggregation)하여 예측합니다.
- 풀링(Pooling) ㅣ 각각의 개별 패치에서 예측된 결과를 집계하는 기술입니다.
- 최대 풀링(Max Pooling) ㅣ 만약 어떤 패치가 질병 존재를 예측하면, 전체 슬라이드도 질병이 있다고 라벨링 됩니다.
- 인스턴스 할당(Instance Assignment) ㅣ 전체 슬라이드에 대해 ‘병변/암’에 대한 라벨은 있지만, 세부적인 패치에서 병변의 위치에 대한 구체적인 정보가 없는 경우 유용합니다.
AI Task
패치 분류(Patch-level Classification)
- WSI의 작은 부분을 분석하여 해당 패치가 질병을 포함하고 있는지를 판단합니다.
- 패치 간의 상관관계나 위치 정보를 사용하지 않고 개별 패치만을 분석합니다.
- 모델은 원본 WSI에서의 위치 관계를 고려하지 않기 때문에 공간 정보가 소실될 수 있습니다.
- 그러나 이 방식은 패치 이미지의 다양성을 확보하고 예측 성능을 향상시킬 수 있습니다.
슬라이드 분류(Slide-level Classification)
- 패치별로 분류된 예측 결과를 종합(Aggregation)하여 전체 슬라이드의 예측을 결정하는 방법입니다.
- 각 패치에 대하여 병변/암 확률을 나타내는 히트맵을 생성할 수 있습니다.
- 슬라이드 자체를 입력으로하여 모델을 학습하기 보다는, 슬라이드에서 추출할 수 있는 여러 특징들을 입력으로 활용합니다.
- AI 입력 특징(Input Features)
- 종양 확률 히트맵(Tumor Probability Heatmaps)에서 파생된 여러 특징들이 입력으로 사용될 수 있습니다.
- 종양 비율(Tumor Percentage) ㅣ 슬라이드 내에서 종양/병변으로 예측된 조직/세포의 비율입니다.
- 종양 영역(Tumor Regions)
- 종양 영역의 마스크를 생성하고, 갯수를 계산합니다.
- 연결된 종양 패치의 최대 크기를 픽셀 단위로 측정하여 가장 큰 종양 영역의 크기를 계산합니다.
- 통계적 특징(Statistical Features) ㅣ 확률 값을 긍정적(종양)과 부정적(정상)으로 분류하여, 통계적 특징을 계산할 수 있습니다.
- 평균(Mean), 중앙값(Median), 최빈값(Mode), 분산(Variance), 표준편차(SD), 최소값(Minimum), 최대값(Maximum), 범위(Range), 총합(Sum)
- AI 입력 특징(Input Features)
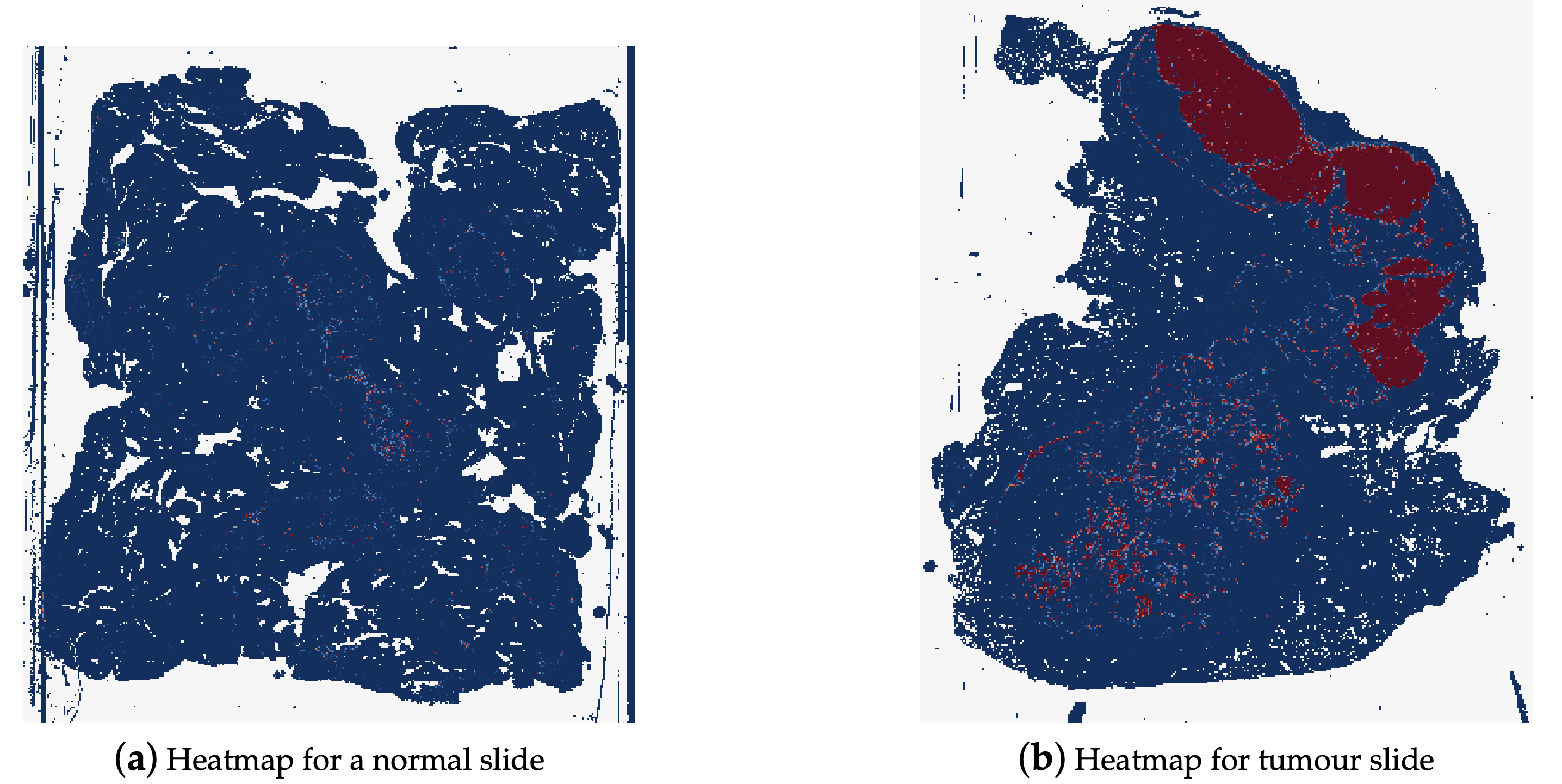 Example of a heatmap for a normal whole slide image and for a tumour whole slide image 1
Example of a heatmap for a normal whole slide image and for a tumour whole slide image 1
WSI와 Foundation Model
- 최근에는 WSI 데이터에 대한 Foundation Model을 개발하려는 연구가 활발하게 진행되고 있습니다.
- Foundation Model(FM)은 대규모 데이터에서 Pre-training이 완료된 모델로, 다양한 Downstream Task에 쉽게 적용할 수 있는 범용 모델을 뜻합니다.
- 기존의 Patch 기반 방법론들과 다르게 WSI에 대한 Foundation Model은 WSI 전체를 이해할 수 있도록 설계되고 있습니다.
- 또한, 의료 WSI 데이터는 수집이나 라벨링의 비용이 크기 때문에, Foundation Model을 활용하여 Self/Semi-supervised Learning 기반 모델을 개발하면 적은 데이터에서도 더 좋은 성능을 기대할 수 있습니다.
- 사례
- Deng et al. (2023) 3 ㅣ 이미지 Segmentation Task의 FM인 SAM(Segment Anything Model) 모델을 병리 WSI에 적용하고, Zero-shot 성능을 세분화하여 평가했습니다.
- Vorontsov et al. (2023) 4 ㅣ 6억개 이상의 파라미터를 가진 ViT 구조를 기반으로 150만장의 H&E염색 WSI로 학습된 FM인 Virchow를 제안했습니다.
- Wang et al. (2024) 5ㅣ CHIEF(Clinical Histopathology Imaging Evaluation Foundation)라고 하는 Weakly-supervised Learning기반 FM 모델을 개발하여 6만장 이상의 WSI 데이터를 학습시켰습니다.
WSI 분석 체크리스트
- 이와 같이 WSI 데이터의 여러 특성으로 인해, 다른 이미지 데이터에 비해 전처리 과정과 AI를 적용하는 방식이 복잡합니다.
- 슬라이드의 품질이나 염색 방식, 배경의 노이즈 등 여러 요소가 AI 분석의 정확성을 해칠 수 있습니다.
- 또한 WSI에서 병변을 정확히 감지하기 위해서는 다양한 수준(Level)의 라벨과 데이터 전처리가 필요합니다.
- 따라서 WSI 분석에는 많은 계산 과학적인 처리가 필요하고 이를 체계적으로 정리한다면 아래와 같이 요약해볼 수 있습니다.
- 하드웨어 및 소프트웨어 ㅣ 학습과 테스트에 사용한 시스템의 하드웨어와 소프트웨어를 문서화합니다.
- 데이터 출처 ㅣ 데이터의 출처와 WSI 데이터에 접근하는 방법을 명확히 기재합니다.
- 데이터 분할(Data Splitting) ㅣ 데이터를 학습, 검증, 테스트 집합으로 어떻게 나누었는지 설명합니다.
- 정규화(Normalization) ㅣ 슬라이드가 색상과 대비에 맞게 정규화 되었는지 설명합니다.
- 배경 제거(Background Removal) ㅣ 슬라이드에서 배경 노이즈와 Artifcat를 어떻게 제거했는지 설명합니다.
- 패치 추출(Patch Extraction) ㅣ 슬라이드에서 패치를 어떻게 추출했는지와 데이터 증강에 대해서 서술합니다.
- 패치 라벨링(Patch-level Annotation/Labeling) ㅣ 패치가 질병에 대한 카테고리나 중증도로 어떻게 라벨링 되었는지를 명시합니다.
- 패치 분류기(Patch-level Classifier) ㅣ 패치 분류기를 어떻게 학습시켰는지(전처리, 방법론, 모델 구조, 하이퍼파라미터)를 설명합니다.
- 슬라이드 분류기(Slide-level Classifier) ㅣ WSI를 분류하는 분류기를 어떻게 학습시켰는지(전처리, 방법론, Aggregation, 모델 구조, 하이퍼파라미터)를 설명합니다.
- 병변 감지 ㅣ 이미지 내에서 병변이 어떻게 감지되었는지를 설명합니다.
- 성능 지표 ㅣ 관련된 모든 성능 지표를 나열합니다.
Conclusion
- 의료 분야에서의 컴퓨터 비전 기술은 꾸준히 발전하고 있으며, 특히 병리학에서 WSI 분석 기술과 AI를 활용한 암 진단 기술들은 조직병리학 또는 세포병리학에서 진단 정확성을 높이고 있습니다.
- WSI는 고해상도 디지털 이미지를 뜻하며 수억 개의 픽셀로 이루어져 있어, 패치 추출(Patch Extraction)과 같은 전처리를 통해 AI 분석의 입력으로 활용됩니다.
- 패치화된 병리 이미지를 CNN기반 모델을 통해 학습하거나, 해당 학습결과와 Slide-level의 피쳐들을 활용한 Patch Aggregation 기술도 함께 연구되고 있습니다.
- 최근에는 기존의 Foundation Model들을 디지털 병리학에 적용하려는 시도가 많이 이루어지고 있습니다.
- 하지만 여전히 WSI 데이터 특성에 따른 기본적인 한계점이 존재합니다.
- 대량의 데이터 처리 ㅣ WSI는 고해상도 거대 이미지이기 때문에 전통적인 AI기법으로 분석하기 어렵습니다.
- 일반적으로 작은 패치로 나누어 분석하는 방법이 사용되지만, 이 과정에서 세부 정보가 손실될 수 있습니다.
- 주석 데이터의 부족 ㅣ WSI를 입력으로하여 AI 모델을 학습하기 위해서는 많은 양의 라벨이 달린 학습 데이터가 필요합니다.
- 의료 이미지 라벨링 과정은 시간이 많이 소모되며, 이로 인해 모델의 학습 효율이 떨어질 수 있습니다.
- 표준화 부족 ㅣ 각 병리학자가 수행하는 분석 방식의 차이는 진단 결과의 일관성을 해칠 수 있습니다.
- WSI 기술을 통한 분석 과정에서 표준화된 진단 프로토콜이 필요해지고 있습니다.
- 대량의 데이터 처리 ㅣ WSI는 고해상도 거대 이미지이기 때문에 전통적인 AI기법으로 분석하기 어렵습니다.
References
Jenkinson, Eleanor, and Ognjen Arandjelović. “Whole slide image understanding in pathology: what is the salient scale of analysis?.” BioMedInformatics 4.1 (2024): 489-518. ↩︎ ↩︎2 ↩︎3
Deng, Ruining, et al. “Segment anything model (sam) for digital pathology: Assess zero-shot segmentation on whole slide imaging.” arXiv preprint arXiv:2304.04155 (2023). ↩︎
Vorontsov, Eugene, et al. “Virchow: A million-slide digital pathology foundation model.” arXiv preprint arXiv:2309.07778 (2023). ↩︎
Wang, Xiyue, et al. “A pathology foundation model for cancer diagnosis and prognosis prediction.” Nature 634.8035 (2024): 970-978. ↩︎